Secuenciación genómica
¿Qué es la secuenciación genómica?
La secuenciación de ADN genómico es un método avanzado que emplea estrategias de secuenciación de alto rendimiento para investigar de manera exhaustiva la totalidad del ADN genómico de un organismo. El objetivo central de este enfoque científico es alcanzar una comprensión profunda de la información genética contenida en el organismo. Esta técnica reviste una importancia excepcional en la investigación biológica, ya que proporciona una base sólida de datos para analizar la compleja relación entre genes, enfermedades y fenotipos. La aplicación de la secuenciación de ADN genómico amplía nuestra percepción sobre las funciones biológicas de un organismo, los mecanismos subyacentes en la patogénesis de enfermedades y la evolución biológica a lo largo del tiempo.
¿Qué es el método de secuenciación de ADN genómico?
Actualmente, las tecnologías de secuenciación de ADN pueden clasificarse en tres categorías principales. La primera comprende la tecnología de secuenciación Sanger, también conocida como secuenciación de primera generación, ampliamente reconocida como el estándar de oro en el sector del diagnóstico clínico. La segunda categoría integra las tecnologías de secuenciación de alto rendimiento (HTS) o de nueva generación (NGS), que han demostrado ser capaces de procesar de manera rápida un enorme volumen de moléculas de ADN con una eficiencia sin precedentes. La tercera categoría abarca las tecnologías de secuenciación de molécula única, que, al no depender de la amplificación por PCR, pueden secuenciar directamente moléculas individuales de ADN. Esta última categoría suele denominarse tecnología de secuenciación de tercera generación.
🔹 Secuenciación Sanger
El método de secuenciación Sanger, técnica arquetípica en la secuenciación de ADN, se basa en la unión específica entre cebadores y las moléculas de ADN molde. Durante el proceso de secuenciación, la ADN polimerasa cataliza la incorporación progresiva de los cuatro tipos de desoxirribonucleótidos trifosfato (dNTP) sobre el ADN molde unido al cebador. La síntesis de nuevas cadenas de ADN se lleva a cabo mediante la formación de enlaces covalentes entre el carbono 3’ de una desoxirribosa y el carbono 5’ del nucleótido siguiente. Este proceso continúa hasta que se incorpora un terminador (ddNTP), que carece de un átomo de oxígeno en el extremo 3’, provocando la detención de la síntesis de la cadena de ADN.
🔹 NGS (Secuenciación de Nueva Generación)
En comparación con el método Sanger, las tecnologías de secuenciación de alto rendimiento, como la plataforma Illumina, ofrecen una eficiencia, capacidad y rentabilidad significativamente superiores. Actualmente, la NGS se ha consolidado como un método ampliamente utilizado en la investigación genómica moderna, con aplicaciones en diversos campos.
Gracias a la tecnología NGS, es posible ejecutar simultáneamente millones de reacciones de secuenciación, lo que constituye un notable avance técnico. Anteriormente, la obtención de secuencias fiables requería la operación coordinada de múltiples mezclas de reacción. Ahora, la información sobre la secuencia de bases puede identificarse directamente durante el proceso sincronizado de extensión y detección de la secuencia.
Con la llegada de la NGS, el alcance de la genómica se ha ampliado considerablemente. En la actualidad, la secuenciación de ADN se ha convertido en un componente esencial en áreas como la ciencia básica, la investigación traslacional, el diagnóstico médico y la ciencia forense. Aunque la tecnología NGS ha logrado importantes avances en la reducción de costos y tiempos, su limitada “longitud de lectura” genera elevados requisitos computacionales para el posterior ensamblaje de genomas. No obstante, se espera que, con el continuo progreso tecnológico y la optimización de los procesos, estos desafíos se superen gradualmente.
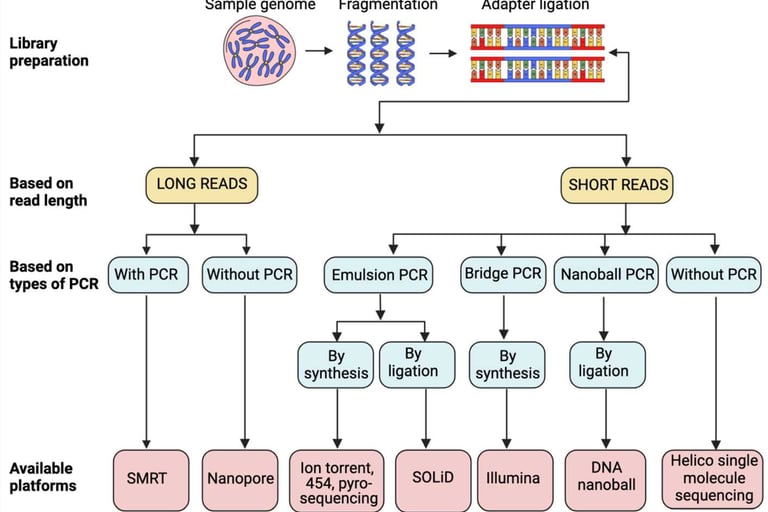
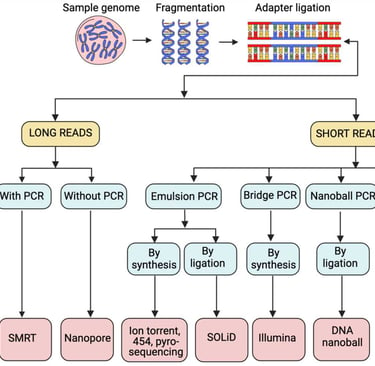
🔹 Secuenciación de Molécula Única
La secuenciación de molécula única, también conocida como secuenciación de lectura larga, está ganando cada vez más atención en la comunidad científica debido a su superioridad en la obtención de lecturas de secuencias extensas. Este tipo de tecnologías se divide principalmente en dos categorías: Secuenciación en Tiempo Real de Molécula Única (SMRT) y secuenciación por nanoporos.
✅ Nuestros Servicios de Secuenciación de ADN
Gracias a plataformas NGS de última generación, tecnologías de vanguardia y un equipo de científicos especializados, BIOGENOMICS ofrece una amplia gama de soluciones genómicas diseñadas para adaptarse a sus diversos objetivos de investigación y presupuestos.
Secuenciamiento de genoma completo
La secuenciación de genoma completo (WGS), una técnica revolucionaria, se ha convertido en una herramienta ampliamente utilizada en la investigación del genoma humano. Su objetivo fundamental es examinar y ordenar de manera exhaustiva la secuencia completa del genoma dentro de una célula biológica, capturando meticulosamente todos los tipos de mutaciones desde la primera hasta la última base de ADN. Esto tiene implicaciones significativas para profundizar en la comprensión de la información genética de un organismo, los mecanismos de las enfermedades y las relaciones entre genes y características fenotípicas.
El desarrollo y la aplicación de la secuenciación de genoma completo se extienden más allá del ser humano, abarcando otros sectores biológicos. En organismos que carecen de genomas de referencia adecuados o que disponen de referencias de baja calidad, las técnicas de de novo sequencing y ensamblaje resultan especialmente valiosas. A través de WGS, los investigadores pueden obtener la información completa del genoma de un organismo, proporcionando una base esencial para explorar funciones génicas, la evolución del genoma, redes de regulación génica y mucho más.
En aplicaciones prácticas, la WGS ha logrado reconocimientos a nivel mundial. Por ejemplo, ha permitido descifrar con éxito las secuencias genómicas de diversas especies animales y vegetales, brindando un sólido respaldo a investigaciones en campos como la agricultura y la medicina. Además, la WGS desempeña un papel crucial en la detección de microorganismos patógenos, la genética forense y los estudios de biodiversidad, entre otros ámbitos.
Secuenciamiento de ADN dirigido
La resecuenciación dirigida es una técnica que se basa principalmente en la secuenciación de amplicones multiplex y la captura híbrida para aislar genes específicos o regiones genómicas de interés y proceder a su secuenciación.
En comparación con la secuenciación de genoma completo (WGS) y la secuenciación de exoma completo (WES), la reselecuenciación dirigida ofrece las siguientes ventajas:
✅ Permite una secuenciación de alta precisión de genes clave, alcanzando profundidades superiores a 500x, lo que facilita la identificación exacta de variantes raras.
✅ Es económicamente eficiente, lo que la convierte en una herramienta ideal para el estudio de genes asociados a enfermedades.
✅ Es capaz de detectar variantes con frecuencias alélicas tan bajas como el 5%.
✅ Permite la identificación fiable de mutaciones hereditarias en una única detección.
Secuenciamiento de ADN mitocondrial
El ADN mitocondrial (mtDNA) es una molécula ubicada en el citoplasma celular, cuya estructura, localización y cantidad influyen directamente en las funciones fisiológicas y en el destino de la célula viva. La tecnología de secuenciación de ADN mitocondrial representa una biotecnología revolucionaria, diseñada específicamente para analizar y secuenciar el mtDNA, el orgánulo clave responsable del metabolismo energético dentro de la célula.
Al ofrecer un mapa detallado de la composición del ADN celular, esta tecnología permite una cuantificación y análisis precisos de los atributos estructurales del ADN. Así, proporciona un retrato completo de las características fenotípicas celulares y abre nuevas vías de investigación para la comunidad científica. Esta técnica ofrece a los investigadores un medio rápido y exacto para explorar en profundidad la estructura y funcionalidad del ADN dentro de la célula. Más aún, desempeña un papel esencial en el esclarecimiento de los misterios relacionados con la estructura genética de las especies y sus respuestas a las variaciones ambientales.
Por lo tanto, la secuenciación de ADN mitocondrial no solo brinda una metodología de investigación más eficaz, sino que también se consolida como una valiosa herramienta de referencia en el ámbito de la salud humana y la investigación médica.
Secuenciamiento de ADN del cloroplasto
Los cloroplastos son uno de los orgánulos más importantes y abundantes en las células vegetales, actuando como el sitio central de la fotosíntesis. La información sobre la estructura y la secuencia de los genomas de cloroplasto tiene un valor significativo para revelar los orígenes, los cambios evolutivos y las relaciones filogenéticas entre diferentes especies. Al mismo tiempo, la tecnología de transformación de cloroplastos muestra un enorme potencial en la mejora genética y la producción de compuestos bioactivos, siendo el análisis estructural y de secuencia del genoma de cloroplasto la base fundamental de este proceso.
Tradicionalmente, la obtención del genoma de cloroplasto de una planta ha implicado el diseño de cebadores degenerados a partir de las secuencias conservadas del genoma cloroplástico, la amplificación de secuencias desconocidas y la amplificación mediante PCR larga. El producto amplificado se somete a secuenciación Sanger, y posteriormente las secuencias se ensamblan para obtener el genoma completo del cloroplasto. Sin embargo, este proceso suele ser laborioso y consumir mucho tiempo.
Con la evolución de las tecnologías científicas y la aparición de nuevas herramientas de secuenciación, numerosos investigadores han mostrado recientemente un creciente interés en la secuenciación de alto rendimiento. En este enfoque, los cloroplastos se aíslan primero, seguidos de la extracción del cpDNA. A partir de genomas cloroplásticos de referencia seleccionados, se utiliza software especializado para ensamblar las secuencias, obteniendo finalmente el genoma completo del cloroplasto. No obstante, este método puede no ser aplicable a todas las especies. Por ejemplo, las hojas de plantas superiores suelen contener grandes cantidades de pigmentos y taninos, lo que dificulta el aislamiento de cloroplastos y la extracción de cpDNA.
Dependiendo de los requisitos específicos de la especie objetivo, se puede optar por la extracción de ADN total o de cpDNA. La información sobre el estatus taxonómico se emplea para buscar secuencias mitocondriales de referencia y diseñar cebadores degenerados que permitan realizar PCR baiting, amplificación de secuencias desconocidas y PCR larga. Finalmente, se obtiene la secuencia completa del genoma cloroplástico mediante una combinación de secuenciación de alto rendimiento y secuenciación Sanger.
Secuenciamiento de amplicones
La secuenciación de amplicones es una técnica poderosa que utiliza cebadores universales específicos para amplificar regiones variables del 16S rDNA, 18S rDNA, ITS o genes funcionales de los microorganismos presentes en diversos entornos. Posteriormente, mediante secuenciación de alto rendimiento, se analizan las variaciones de las secuencias y la información sobre la abundancia de los productos generados por la reacción en cadena de la polimerasa (PCR).
Este enfoque permite investigar la diversidad y los patrones de distribución de las comunidades microbianas en ambientes determinados, revelando la abundancia relativa y las relaciones evolutivas entre la amplia variedad de especies microbianas que habitan en las muestras ambientales.
Secuenciamiento de genoma de virus
La secuenciación de genoma completo de virus (VWGS) consiste en un análisis exhaustivo de las secuencias genómicas de los virus mediante plataformas de segunda y tercera generación. Aprovechando metodologías bioinformáticas, este enfoque permite interpretar la información codificante y realizar estudios profundos sobre los sistemas patogénicos virales y la trayectoria evolutiva de sus genomas. Disciplinas como la genómica estructural y comparativa —incluyendo análisis diferenciales, análisis de genes homólogos, análisis de colinealidad y estudios de evolución de especies— utilizan estas técnicas para examinar detalladamente estos aspectos. Dichos esfuerzos enriquecen nuestra comprensión de la diversidad, ecología, adaptabilidad y patrones evolutivos de los virus, y contribuyen a predecir la aparición de enfermedades infecciosas emergentes.